京コンピュータを用いたウイルスの全原子シミュレーション
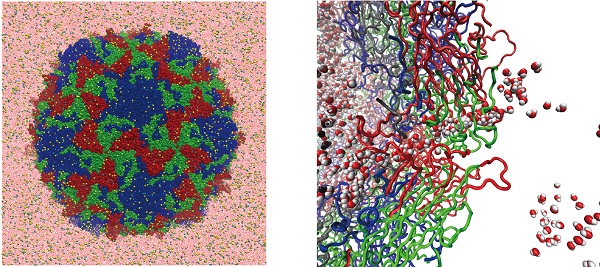
ウイルスはタンパク質により構成された殻(カプシド),殻内部の核酸,ときにエンベロープと呼ばれる脂質二重層膜の外被からなる。筆者らは直径30 nmほどの比較的小さなポリオウイルスエンプティカプシドまるごと1 つを扱った全原子分子動力学(MD)シミュレーションを行うことで,ポリオウイルスに関わる物理化学を分子レベルから明らかにしようとしている。溶媒を含め計算系には,原子が107 個程度含まれる。この規模での長距離静電相互作用を含むMD計算は京コンピュータと汎用MD ソフトMODYLAS1)の組合せにより初めて可能になった。
X 線結晶構造解析により特定されたポリオウイルスの分子構造2)をもとにした温度圧力一定条件下でMD シミュレーションにより,カプシドタンパク質の構造及びダイナミクス,つまり残基の熱揺らぎ,殻全体の伸縮などがはじめて明らかになった。またカプシドを横切る水分子数の解析からカプシドが電解質イオンを透過せず水分子のみを透過する半透膜的性質を持つことを明らかにした。
中性pH 条件下でともに負に帯電したポリオウイルスとその受容体CD155 がなぜ好んで会合するかという謎も,今後のシミュレーションにより解明されるであろう。そして,その研究成果は新しい発想での抗ウイルス剤の開発にもつながると期待される。
1) Y. Andoh et al., J. Chem. Theory Compt. 2013, 9, 3201.
2) S. T. Miller, J. M. Hogle, D. J. Filman, J. Mol. Biol. 2001, 307, 499.
安藤嘉倫 名古屋大学大学院工学研究科